
王艳丽研究组揭示了一种高切割活性的II-D型Cas9的结构与机制
CRISPR-Cas系统是广泛存在于细菌和古菌体内的一种RNA引导的适应性免疫系统。Cas9是II型CRISPR-Cas系统的标志性蛋白,具有HNH和RuvC两个核酸酶结构域,通过与crRNA和tracrRNA或单链sgRNA结合,识别含有PAM序列且与crRNA互补的双链DNA序列,实现靶向切割。化脓链球菌中的Cas9(SpCas9)因其高切割效率和优秀的基因编辑表现而成为目前应用最广泛的Cas9,但其较大的分子量限制了其通过腺相关病毒(AAV)载体进行递送。因此,从自然界挖掘或者通过蛋白质工程获得更小分子量且保持高切割活性的Cas9,成为基因编辑领域的一项重要需求。
2025年8月11日,中国科学院生物物理研究所王艳丽团队在《Nature Communications》杂志在线发表了题为"Structural insights into Type II-D Cas9 and its robust cleavage activity"的研究论文,揭示了一种高切割活性的II-D型Cas9的结构与机制。
一种来源于硝化螺旋菌的II-D型Cas9(NsCas9d),仅由762个氨基酸组成,较常见的II-A/B/C型Cas9更为紧凑(图A)。II-D型Cas9在进化树分析中位于独立的进化枝。研究人员在宏基因组数据中鉴定出了NsCas9d相关的重复序列和间隔序列,并为NsCas9d设计了sgRNA。
体外切割实验表明,底物dsDNA需要与sgRNA形成至少20 nt的配对,NsCas9d才能发挥与SpCas9相当的dsDNA切割活性(图B)。结合PAM depletion实验和高通量测序,研究人员鉴定出NsCas9d识别5′-NRG-3′ PAM序列,R可以是A或G。当PAM是5′-NGG-3′时观察到了最高的体外切割效率(图C)。
相较于先前报道的Cas9d-MG34-1的结构模型,该研究解析了NsCas9d-sgRNA-dsDNA三元复合物的2.86 Å分辨率的冷冻电镜结构,首次报道了II-D型Cas9完整的HNH和RuvC结构域结构(图D)。NsCas9d的sgRNA结构与ωRNA的5′端相近,缺少ωRNA的3′端结构。优化后的sgRNA与经典的Cas9的sgRNA长度相近。在复合物结构中,HNH结构域的催化中心正对着其要进行切割的单链DNA,说明NsCas9d处于催化活性状态。
在复合物结构模型中,dsRNA的PAM序列位于PI和WED结构域形成的带有正电荷的结合通道。NsCas9d切割产物是有3-nt 5′-overhang的粘性末端,区别于典型的Cas9切割产生的平末端,而与Cas12的切割产物类似(图E)。这一特征在进行基因插入等基因编辑操作时有利于提高DNA修复过程的效率和可预测性。
为了验证NsCas9d成为哺乳动物细胞基因编辑工具的潜力,研究人员在HEK293T细胞中对NsCas9d和SpCas9的体内编辑效率进行了比较。NsCas9d能够成功靶向目的序列,发挥基因编辑工具的作用,但是编辑效率明显低于SpCas9。针对NsCas9d进行结构性和技术性的优化有望提高编辑效率,使得NsCas9d满足基因编辑领域对更小分子量且保持高切割活性的Cas9需求。
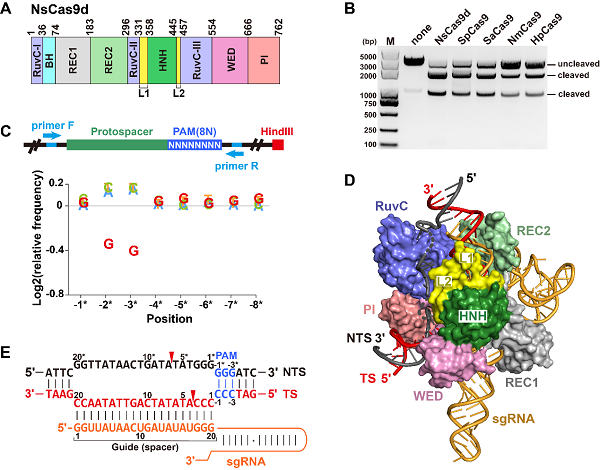
图:NsCas9d的切割活性与三元复合物结构模型
中国科学院生物物理研究所王艳丽研究员为文章的通讯作者,博士研究生王康康为第一作者。该研究得到了国家重点研发计划、北京市科学技术委员会、国家自然科学基金、北京市自然科学基金、中国科学院的项目资助。生物物理所生物成像中心为电镜数据收集提供了技术支持。
文章链接:https://doi.org/10.1038/s41467-025-62128-8
(供稿:王艳丽研究组)
附件下载: